Rafael A. Rosales
Introdução a Probabilidade e Estatística II (MAN) 2024
Prova de recuperação: 27/01/2026
lugar: sala 600B DCM, hora: 10h-12h.
O Jupiterweb entre outras informações fornece o conteudo desta disciplina.
Caso tiverem dúvidas sobre alguns dos exercicios ou sobre o envio de algum trabalho via e-nail, revissão de prova, etc, escrevam ao e-mail
prob2.man2025@gmail.com
Não escrevam para o meu e-mail @usp (sua mensagem será tratada como spam).
Avaliação
- Prova 1: 6 de outubro, das 8:00 as 10:00. prova.pdf, respostas.pdf
- Prova 2: 1 de dezembro, das 8:00 as 10:00.
- recuperação: data/hora/lugar serão anunciados neste site
Nota: (P1+P2)/2. (Jupiterweb)
Lista de exercícios/Gabaritos
Todos os exercícios e alguns tópicos adicionais podem ser encontrados na seguinte lista.
O gabarito contem as respostas para algumas das questões da lista. Sugiro fortemente tentar resolver as questões de maneira independente, antes de ver o gabarito.
Dados/R
Os dados a serem utilizados na lista de exercícios e no decorrer do semestre são os seguintes:
orto,
escola,
cancer,
energy,
chicken,
stroke,
cabbage,
Cars93,
hospital,
trabalho,
e
Covid-RP. Qualquer um destes dados pode ser carregado em R utilizando a função read.table, por exemplo
dados <- read.table("http://dcm.ffclrp.usp.br/~rrosales/aulas/cancer.txt", head=TRUE)
R é livre e pode ser baixado do seguinte site. Instalar R é relativamente simples. O site oficial também inclui vários tipos de documentação, inclusive em portugês (no menu a esqueda no site oficial: Contributed). O seguinte documento apresenta uma introdução mínima a linguagem R.
O primeiro Capítulo da referência 1, listada na Bibliografía abaixo, pode ser obtido aqui.
Bibliografía
- Marcos Nascimento Magalhães. Noções de probabilidade e estatística. Edusp 2004/05.
- Wilton de O. Bussab, Pedro A. Morettin. Estatística Básica. Saraiva, 2004.
- Heleno Bolfarine, Mónica Carneiro Sandoval. Introdução a Inferência Estatítica. SBM, 2010.
R na sala de aula: simulações, análises, etc
05/08/25
Amostras/estimadores 1: média amostral
Os seguintes comandos tem por objetivo ilustrar a definição de amostras e estimadores.
A função sample pode ser
utilizada para gerar amostras de uma
população descrita por uma variável
aleatória discreta com um número finito de
valores. Por exemplo,
sample(x=c(1,2,3,4,5,6), size=300, replace=TRUE, prob=c(2/3,1/15,1/15,1/15,1/15,1/15))
gera a amostra
[1] 1 1 1 1 3 1 1 1 1 6 6 1 1 1 1 5 1 6 1 6 4 4 1 1 1 1 6 1 1 2 1 1 1 1 1 1 1 [38] 1 1 3 1 1 3 1 4 1 4 4 1 5 1 1 1 1 5 1 1 4 1 1 1 1 1 3 1 4 1 5 1 1 1 5 1 1 [75] 1 1 4 1 1 6 1 1 5 2 1 6 1 1 1 1 1 1 1 4 1 3 4 1 1 5 2 4 2 1 1 1 1 2 4 1 1 [112] 2 1 1 1 1 3 4 1 1 1 3 5 5 2 1 1 5 3 4 1 3 1 5 1 2 1 1 1 1 6 2 1 1 1 1 3 5 [149] 1 2 5 1 4 2 4 1 1 1 3 1 4 1 1 1 1 1 4 1 1 2 5 1 1 4 1 1 1 1 1 3 1 1 1 3 3 [186] 1 1 3 1 1 1 4 3 1 3 1 6 1 1 1 4 5 1 1 1 2 4 1 1 1 4 1 6 5 1 1 1 6 1 2 3 1 [223] 5 1 1 4 5 3 1 1 4 1 1 1 5 1 2 1 1 1 1 1 6 6 6 3 4 2 2 1 1 1 1 1 4 1 2 5 1 [260] 1 6 1 1 1 4 4 1 1 1 1 1 6 3 2 3 5 1 4 1 1 1 1 1 3 1 4 1 1 2 4 1 1 1 1 1 4 [297] 2 1 1 1
a qual representa o resultado de termos lançado 300 vezes um dado não honesto, com probabilidade 2/3 de resultar em 1. Para simular 10 lançamentos de uma moeda honesta e de outra com probabilidade 1/4 de resultar em cara podemos fazer (1 representa cara)
sample(x=c(0,1), size=10, replace=TRUE, prob=c(1/2,1/2)) sample(x=c(0,1), size=10, replace=TRUE, prob=c(3/4,1/4))
R posue funções para gerar amostras de uma grande quantidade de distribuições, alguns exemplos são: rnorm, rpois, rgeom, rbinom, rchisq e rt. O seguinte exemplo gera uma amostra de tamanho 200 de uma população Poisson$(\lambda)$ com $\lambda=10$,
rpois(lambda=10, n=200) [1] 12 15 11 10 10 12 12 7 7 10 14 6 15 11 9 9 6 14 6 9 15 15 8 13 6 [26] 13 12 10 11 8 12 11 9 10 10 8 9 13 4 7 9 9 18 13 10 15 8 11 7 10 [51] 10 9 15 5 10 13 4 7 9 10 10 12 8 12 7 15 14 11 6 14 9 5 12 12 15 [76] 15 14 12 13 11 7 12 6 10 12 3 8 6 13 5 14 9 12 11 12 6 8 5 11 17 [101] 3 14 11 12 10 11 6 9 8 8 2 9 6 14 13 7 12 10 10 9 9 11 11 8 10 [126] 9 5 13 17 13 7 9 5 8 17 13 10 11 8 7 9 10 8 4 8 7 6 11 10 14 [151] 12 11 15 8 10 8 5 8 12 6 11 10 21 6 11 10 4 8 9 12 7 14 11 11 20 [176] 10 12 14 6 13 10 8 7 6 17 5 9 5 6 5 10 10 14 15 9 6 9 12 9 8
Suponhamos que a polulação das alturas dos estudantes da USP seja normal com média 1.68 (m) e variância 0.01. A seguinte linha utiliza a função rnorm para gerar uma amostra de tamanho 10 desta população e armacena os valores gerados no vetor alturas,
alturas <- rnorm(n=10, mean=1.68, sd = sqrt(0.01))
Definimos a seguir uma função da amostra, $g(X_1, \ldots, X_n)$, com o objetivo de estimar a média da população (suponha que o valor 1.68 utilizado para gerar a mostra seja realmente um parâmetro desconhecido),
g <- function(x){
print(sum(x)/length(x))
}
A função $g$ calcula a media amostral, isto é, para a amostra $x_1, x_2, \ldots, x_{10}$, o estatístico $g$ retorna \[g(x_1, \ldots, x_{10}) = \frac{1}{10}\sum_{i=1}^{10} x_i.\] Em particular, lembramos que este é o valor numérico do estimador $\bar X = \frac{1}{n} \sum_{i=1}^n X_i$ na amostra \[(X_1, X_2, \ldots, X_n)(\omega) = (x_1, x_2, \ldots, x_n).\] No R, o resultado ao aplicarmos o estimador na amostra gerada é
g(alturas)
Podemos gerar mais amostras e observar os valores do estimador em cada uma, por exemplo
for (i in 1:5) g(rnorm(n=10, mean=1.68, sd = sqrt(0.01))) [1] 1.706853 [1] 1.662526 [1] 1.690708 [1] 1.688903 [1] 1.752636
gera 5 amostras e calcula a media amostral de cada
uma. Em sala de aula denotamos $g(X_1, \ldots, X_n)$ por
$\bar X$, o estimador média amostral. O importante
aqui é observarmos como a depender da amostra podem
resultar valores diferentes para o estimador $\bar X$: estes
valoressão todos estimativas de $\bar X$.
Cada chamada a rnorm muda a semente utilizada
pelo gerador de números aleatórios utilizados
por esta função gerando portanto amostras
possívelmente diferentes. Em lugar
de g(), definida acima, R possue a
função mean.
05/08/25
estimadores 2: variância amostral
Dada uma amostra $X_1$, $X_2$, $\ldots$, $X_n$ de uma população $X$, seja $S^2$ a variável aleatória definida por \[ S^2 = \frac{1}{n-1}\sum_{i=1}^n (X_i -\bar X)^2. \] $S^2$, conhecido como a variância amostral, é utilizado como um estimador da variância da população $\sigma^2 =$Var$(X)$. $S^2$ pode ser implementado pela função,
S2 <- function(x){
n <- length(x)
xbarra <- sum(x)/n
print(sum((x-xbarra)^2/(n-1)))
}
Assim, para $\sigma = 0.1 = \sqrt{0.01}$ e 5 amostras de tamanho 10
temos
for (i in 1:5) S2(rnorm(n=10, mean=1.68, sd = sqrt(0.01))) [1] 0.005877579 [1] 0.01414221 [1] 0.007516044 [1] 0.007520869 [1] 0.01134922oDe novo, o importante aqui é vocês observar como o valor de $S^2$ varia aleatóriamente de amostra em amostra.
No R, as funções g() e S2() são
implementadas respectivamente por mean()
e var(); logo não é necessário definilas, mas o
meu objetivo era explicar como isto pode ser feito.
11/08/25
estimadores 3: distribuição empírica
O script empirical_pdf.R tem por objetivo mostrar a performance do estimador da funcão de distribuicão de X conhecido como a distribuição empirica, isto é do estimador \[ \widehat F_{X_1, \ldots, X_n}(x) = \frac{1}{n} \sum_{i=1}^n 1_{\{X_i \leq x\}} \] para a função de distribuição $F(x)$ da população $X$. O exemplo descrito em sala de aula é apresentado a seguir. source("http://dcm.ffclrp.usp.br/~rrosales/aulas/empirical_pdf.R")
dado(N=10)
plotFn(dado(N=10))
for (i in 1:6) plotFn(dado(N=10), add=T)
plotFn(dado(N=10000), add=T, col.hor="red", lwd=3)
# amostras de uma populacao normal padrao (mu=0, sd=1)
plotFn(normal(N=30), xlim=c(-3,3))
for (i in 1:3) plotFn(normal(N=30), add=T)
plotFn(normal(N=15000), add=T, col.hor="blue", lwd=3)
A primeira linha carrega as funções
definidas em empirical_pdf.R. A
segunda linha gera uma amostra $X_1$, $X_2$,
$\ldots$, $X_{10}$, a qual representa o
lançamento de um dado honesto 10
vezes. Isto é implementado com a
função dado, a qual por
sua vez utiliza a função do
R sample.
A terceira linha calcula e grafica a estimativa
para $\widehat F_{n}(x)$ baseada na amostra gerada
com dado. A quarta linha calcula e
grafica mais outras seis estimativas, cada uma
obtida com uma amostra diferente (subsequentes
chamadas a sample geram amostras
"aleatórias" diferentes; tente você
mesmo). A quinta linha calcula e grafica a
estimativa obtida com uma amostra de tamanho
n=10000, ou seja uma sequência de
números obtida ao lançarmos 10000 um
dado!. A estimativa desta ultima deve estar bem
próxima da função de
distribuição de um dado honesto.
As ultimas linhas repetem esta experiencia para
amostras de uma população normal
padrão, isto é, com média 0 e
variância 1. Ambos os resultados são
mostrados na figura abaixo.
12/08/25
Aula de R
Todos os comando utilizados durante a aula de R podem ser acesados no seguinte arquivo.
30/08/22: Lei (fraca) dos Grandes Números
As simulações apresentadas na primeira aula para ilustrar um exemplo da Lei dos Grandes Números, de fato uma simulação da primeira Lei dos Grandes Números devida a J. Bernoulli, podem ser reproduzidas da seguinte forma. Inicie R e digite
source("http://dcm.ffclrp.usp.br/~rrosales/aulas/moedaWLLN.R")
Este script define a
função moedaWLLN(), a qual pode
ser utilizada para simular o lançamento de uma
moeda com prob. $p$ de sair cara N
vezes. MoedaWLLN() aceita os seguintes
argumentos além de N e
$p$: m o número de moedas a serem
lançadas e coins uma variável
com dois valores TRUE/FALSE. Digite o
seguinte e observe o resultado de cada linha
moedaWLLN(m=1, p=0.25) moedaWLLN(m=1, N=300, p=0.25) moedaWLLN(m=1, N=3000, p=0.25) moedaWLLN(m=1, N=30000, p=0.25) moedaWLLN(m=15, N=3000, coins=FALSE, p=0.5)
Cada uma da linhas acima gera um gráfico da
fração reativa de caras $S_N/N$ em
função de $N$. $S_N$ corresponde a soma dos
resultados de $N$ variáveis aleatórias
Bernoulli $S_N = \xi_1 + \xi_2 + \ldots + \xi_N$. A ultima
linha fornece um grafico de 15 trajetorias de $S_N/N$
obtidas ao lançar a mesma moeda $N$ vezes um total
de 15 vezes. Isto ultimo permite visualizar a
noção de convergência em probabilidade
\[
\frac{S_N(\omega)}{N} \underset{\mathbb{P}}{\longrightarrow} p,
\]
ou seja, para qualquer $\epsilon > 0$,
\[
\lim_{N \to \infty} \mathbb{P}\bigg(\bigg\{\omega \in \Omega\ :\ \bigg|\frac{S_N(\omega)}{N} - p\bigg| > \epsilon\bigg\}\bigg) = 0 .
\]
Cada caminho na simulação corresponde a um evento elementar $\omega$, determinado por uma sequencia específica de caras e coroas.
É possível visualizar o corpo da função moedaWLLN, simplesmentes ao digitar
moedaWLLN
09/09/25
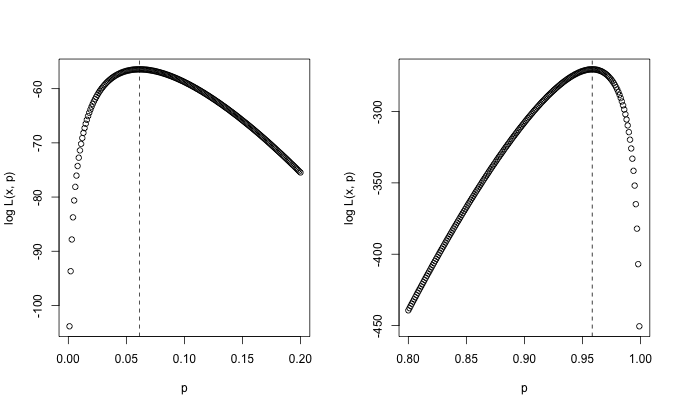
máxima verosimilhança
amostra <- rgeom(15, 1/10)
p.maxver <- length(amostra)/(sum(amostra)+length(amostra))
LogVerGeom <- function(x=amostra, p=1/2) {
length(x)*log(p) + sum(x)*log(1-p)
}
p <- seq(0,0.2,0.001)
plot(p, LogVerGeom(amostra, p))
abline(v=p.maxver, lty=2)
O código acima fornece um exemplo do método
máxima verossimilhança para uma
população $X$ com distribuiça;ão
Geometrica($p$), $p$=1/10. Suponha que $X$ representa o tempo
de espera em um ponto de onibus qualquer em Ribeirão
Preto; $p$ representa a probabilidade de pegarmos o onibus em
um minuto. Se $p$=1/10, em méia devemos esperar 10
minutos. A primeira linha do código acima gera uma
amostra de tamanho 15 da população. R ao
contrario do feito em aula parameterisa o modelo Geometrico da
seguinte forma \[P(X = k) = (1-p)^k p, \qquad k = 0, 1,
\ldots\] Neste caso o estimador de máxima
verossimilhança é $\hat{p}_{MV} = n/\sum_i
(X_i-1)$, sendo $X_i$, $1 \leq i \leq n$ a amostra de $X$. A
primiera linha do código gera uma amostra com $n=15$. A
segunda linha calcula a estimativa do estimador
$\hat{p}_{MV}$. A terceira linha define a
função LogVerGeom, a qual calcula o
logaritmo da função de
verosimilhançã na amostra, dado um valor para
$p$. A quarta linha define um vetor com possíveis
valores para $p$. A quinta linha grafica o logaritmo da
verosimilhança em funçao de $p$. Por ultimo
adicionamos a posição da estimativa para
$p$. Esta ultima deveria estar no lugar de máxima
verossimilhança. Sugiro voce executar o script acima tal
qual e logo substituindo a primeira e a quarta linha por
amostra <- rgeom(1500, 0.95) p <- seq(0.8,1,0.001) p.maxver <- length(amostra)/(sum(amostra)+length(amostra)) plot(p, LogVerGeom(amostra, p), xlab="p", ylab="log L(x, p)") abline(v=p.maxver, lty=2)
O valor $p$=0.95 poderia ser utilizado para modelar a probabilidade de pegarmos o onibus no primiero minuto na Alemanha, por exemplo. Os graficos gerados pelos comandos acima é apresentado abaixo.
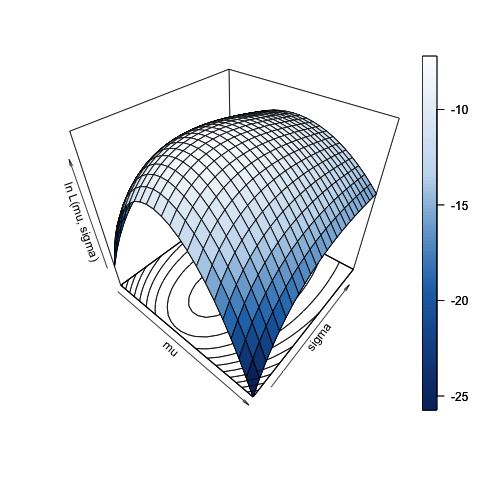
O
script
NormalLik.R simula uma amostra de tamanho 40 de uma
população normal com média 1.68 e
variância 0.1 (com o intuito de simular uma amostra
das alturas de vocés) e posteriormente calcula e
grafica a superficie de verossimilhanca para um
determinado dominio dos valores para a média e a
variância. O grafico abaixo apresenta o
resultado. Tente executar o script várias e observe
a variação da superficie determinada pela
amostragem! Observe que para poder gerar o grafico,
é necessário instalar a
livraria plot3D, digitanto, por exemplo,
desde a linha de comando do
R install.packages("plot3D").
O script também imprime o valor das estimativas para os estimadores de máxima verossimilhança baseados na amostra; veja os numeros impresos na linha de comando que aparecem depois de digitar
source("http://dcm.ffclrp.usp.br/~rrosales/aulas/NormalLik.R")
15/09/21
Teorema Central do Limite
A seguinte linha carrega o script que simula o Teorema Central de Limite para sequências de variáveis aleatórias Bernoulli($p$), i.e. o TCL de De Moivre - Laplace como explicado em sala de aula: em cada iteração é gerada uma amostra de $n$ Bernoullis $\xi_1$, $\ldots$, $\xi_n$ independentes, logo é calculada a média aritmética $S_n/n= \frac{1}{n}\sum_{i=1}^{n} \xi_i$ para ser corretamente padronizada, substraindo a sua média e dividindo pelo seu desvio padrão, \[ \frac{S_n/n - \mathbb E\big[S_n/n\big]}{\text{Var}\big(S_n/n\big)^{1/2}} = \frac{S_n - n p}{p(1-p)\sqrt{n}}. \] Este procedimento é repetido 45 vezes e o histograma dos 45 valores sobtidos é graficado. Este procedimento tudo é repetido várias vezes e o resultado de cada repetição é acumulado no mesmo histograma. O ćodigo deve ser carregado como
source("http://dcm.ffclrp.usp.be/~rrosales/aulas/CLT_a.R")
A simulação ê realizada digitando
animCLT()
A simulação é finalizada ao digitar CTRL-C ou CTRL-Z ou clickando no icone "STOP" no RStudio. Uma vez finalizada a simulação, é possível sobrepor sobre o histograma gerado a densidade normal padrão com
addens(text=TRUE)
16/09/25
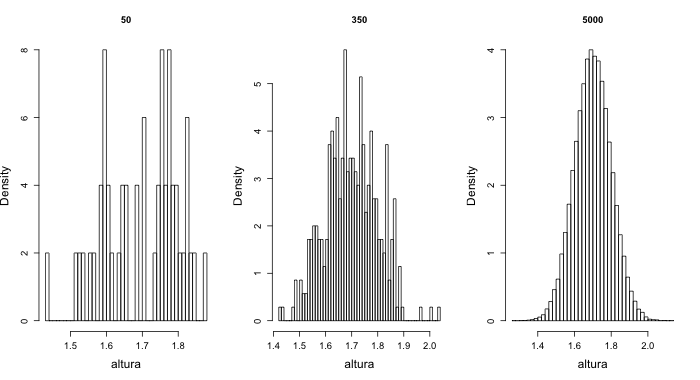
As simulações apresentadas
em aula ilustram a convergência (em probabilidade) do histograma
$\widehat h_{n}(x)$ a distribuição da
população $f$,\[\widehat h_n(x) \underset{\mathbb
P}{\longrightarrow} f(x).\] No código
abaixo, sala.MAN.* é uma amostra de
tamanho *=50, 350, 5000 de uma populacao normal com media
1.7 e desvio padrão 0.1, utilizada para simular a
altura de vocês (em metros). Este exemplo não foi
realizado na aula, mas queiro disponibilizar aqui para
voces terem ainda mais exemplos.
sala.MAN.50 <- rnorm(52, mean=1.7, sd=0.1) hist(sala.MAN.50, breaks=50, main="50") sala.MAN.350 <- rnorm(350, mean=1.7, sd=0.1) hist(sala.MAN.350, breaks=50, main="350") sala.MAN.50000 <- rnorm(50000, mean=1.7, sd=0.1) hist(sala.MAN.50000, breaks=50, main="50000")
O resultado é apresentado na figura abaixo.
O seguinte exemplo, feito em aula, ilustra a convergencia do histograma quando a densidade da população segue o modelo Exponencial($\lambda$) com $\lambda=3$
T.50 <- rexp(n=50, rate=3) hist(T.50, breaks=50, main="50", xlab="tempo") T.350 <- rexp(n=350, rate=3) hist(T.350, breaks=50, main="350", xlab="tempo") T.50000 <- rexp(n=50000, rate=3) hist(T.50000, breaks=50, main="50000", xlab="tempo")
Neste caso $X$ pode representar o tempo de espera para ser atendido em um posto de saude em minutos ($\lambda = 3$ é o tempo médio de espera).
17/11/24 regressão linear aplicada a casos de COVID-19 na região de Ribeirão Preto
regressão linear aplicada a casos de COVID-19 na região de Ribeirão Preto
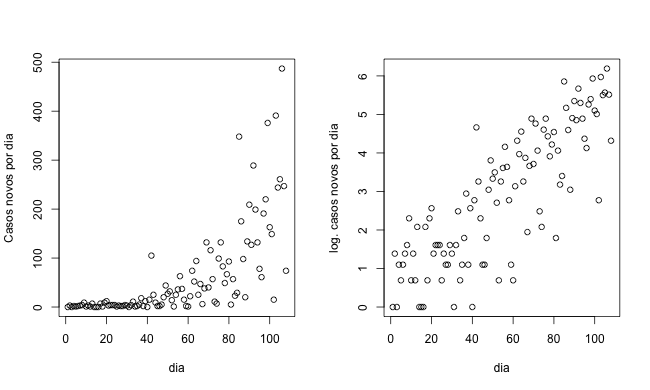
O seguinte codigo ilustra a analise realizada com os dados covid-RP. Os dados apresentam o número de casos novos de COVID-19 reportados por dia na região de Ribeirão Preto desde o 27/03/21 até 12/07/21 (dados oficiais da Secretaria da Saude; mais detalhes podem ser encontrados no cabeçado do próprio arquivo de dados).
Os seguintes comandos importam os dados e geram um gráfico do numero de casos novos por dia e também um gráfico do logaritmo do numero de casos novos.
dados <- read.table("Covid-RP.txt", head=TRUE)
attach(dados)
help(par)
op <- par(mfrow = c(1, 2))
plot(dia, Nov, xlab="dia", ylab="Casos novos por dia")
plot(dia, log(Nov+1), xlab="dia", ylab="log. casos novos por dia")
O resultado é apresentado na seguinte figura.
O seguinte script reliza uma regressão linear aos dados
transformados logaritmicamente. O comando summary
fornece um resumo da analise de regressão; sugiro rever as minhas
duas ultimas aulas e o texto na lista de exercícios relativo a
análise de regressão para entender o output de summary.
regre <- lm(log(Nov+1) ~ dia)
summary(regre)
Call:
lm(formula = log(Nov + 1) ~ dia)
Residuals:
Min 1Q Median 3Q Max
-2.4646 -0.5225 0.1721 0.6833 2.3392
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.379256 0.187963 2.018 0.0461 *
dia 0.046309 0.002994 15.469 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.9699 on 106 degrees of freedom
Multiple R-squared: 0.693, Adjusted R-squared: 0.6901
F-statistic: 239.3 on 1 and 106 DF, p-value: < 2.2e-16
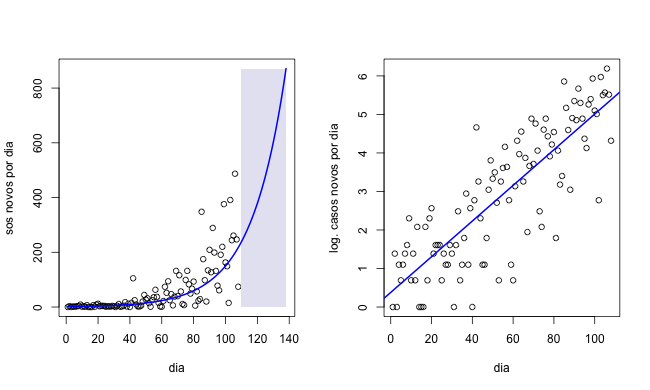
O seguinte código sobrepoe a reta de regressão $y = \hat\beta x + \hat \alpha$ obtida no passo anterior aos dados transformados logaritmicamente e também mostra o grafico da função $e^{\hat\beta x + \hat\alpha}$ sobreposta no dados na escala original.
x <- seq(1,length(dia)+30) beta <- 0.046309 alpha <- 0.379256 f <- exp(x*beta + alpha) par(mfrow = c(1,2)) plot(dia, Nov, xlim=c(1,length(dia)+30), ylim=c(0,max(f)), xlab="dia", ylab="Casos novos por dia") rect(110, 0, 108+30, max(f), col = rgb(0.9,0.9,0.95), border = NA) lines(x, f, lwd=2, col="blue") plot(dia, log(Nov+1),xlab="dia", ylab="log. casos novos por dia") abline(regre, lwd=2, col="blue")
O resultado é apresentado na figura a baixo.
No grafico na escala original é considerada uma projeção do modelo de regressão 30 dias depois do ultimo dado em 12/07 (área achurada em azul). Isto ultimo só faz sentido se supomos que a fase de crescimente exponencial ainda persista nos próximos 30 dias. Não epoca na qual foram coletados estes dados realmente não sabiamos se isto último seria uma boa predição, mas parecia ser apropriado. De qualquer forma o objetivo desta analise era (e ainda é) a necessidade de avaliar de maneira objetiva se o lock down deveria ter sido implementado nesse momento ou não. Felizmente este ultimo foi de fato implementado nos dias seguintes ao último regristro nos dados utilizados. Espero que seja um incentivo para estimular futuros estudos e mostrar que o método de regressão pode ser, quando utilizado corretamente, uma ferramenta poderosa.